La variación en cualquier sistema de características (morfológicas o moleculares) puede reflejar una combinación de los factores ecológicos, genéticos e históricos. Un componente importante de la variación es la que se debe primariamente a diferencias en la constitución genética individual e intrapoblacional. Esta variación inducida genéticamente puede estar asociada a procesos sexuales o no sexuales. La variación genética asociada a procesos no sexuales, por lo general, es continua. Sin embargo, algunas características (morfológicas y moleculares) varían de manera discontinua. Esta variación discreta se denomina polimorfismo. La existencia de polimorfismo en las poblaciones es de gran importancia para el estudio de procesos biológicos en la ecología evolutiva y la sistemática.
Gracias a que existe variación a nivel individual, poblacional y específica, es posible encontrar atributos heredables que hacen posible inferir si los semaforontes o variantes bajo examen pertenecen o no al mismo organismo, a la misma población o a la misma especie. La individualidad de estas tres entidades no siempre es tan evidente y obvia, especialmente al nivel de poblaciones y especies, por lo que se necesitan marcadores cuya presencia común sea indicadora de conexión ontogenética, genética o histórica.
Durante la década pasada las estrategias clásicas para detectar polimorfismo como la morfometría, la citología, la genética y la bioquímica, se han complementado con técnicas moleculares. Con el desarrollo de los llamados marcadores moleculares, se han facilitado las investigaciones en disciplinas como taxonomía, ecología y genética.
Los marcadores moleculares se detectan a través de una serie de métodos que exploran la variación de los organismos a nivel de proteínas o ADN globalmente y a nivel de un locus o varios loci en particular.
A nivel de proteínas los métodos usados son
- las reacciones inmunológicas,
- la electroforesis de isoenzimas y
- la secuencia de aminoácidos de las proteínas.
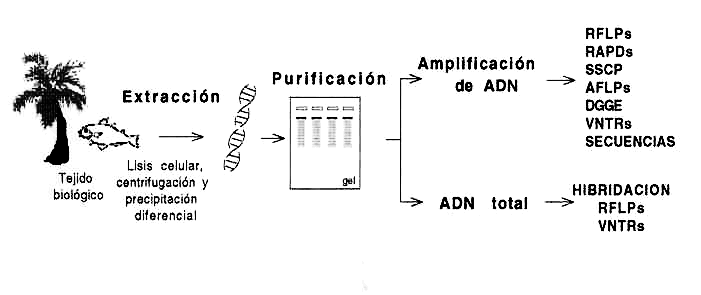
Fig. 1. Extracción de ADN y dos vías alternativas para la búsqueda de marcadores moleculares. El ADN extraído se purifica mediante varios métodos (ultracentrifugación, columnas o geles). Este ADN purificado se recupera para examinar la variación a nivel de todo el genoma o para amplificar una región específica.
A nivel del ADN (Fig. 1) las técnicas mas comunes son:
- la hibridación de ADN,
- los fragmentos de restricción de longitud polimórfica ("RFLPs"),
- los mapas génicos,
- el polimorfismo del ADN amplificado al azar ("RAPDs"),
- la huella digital del ADN ("VNTRs" o "fingerprinting"),
- los microsatélites,
- la conformación polimórfica de cadena sencilla ("SSCP"),
- los mapas de sitios de restricción polimórficos en productos de PCR ("MRSPs"),
- la amplificación selectiva de fragmenos de restricción ("AFLPs"),
- electroforesis de ADN en geles en gradientes desnaturalizantes ("DGGE") y
- la secuencia de ADN, entre otros.
Los métodos que exploran la variación de los organismos a nivel de proteínas o ADN han ayudado a detectar polimorfismo a nivel individual, poblacional o específico. Esto ha favorecido la investigación de problemas ecológicos o sistemáticos como paternidad, diversidad genética, heterocigosidad, variación geográfica, hibridación, delimitación geográfica de especies, especiación y filogenias de especies entre otros.
Aunque es común que en la literatura se promuevan ideas sobre la superioridad de un tipo de marcador sobre otro. La realidad es que todos los tipos de marcadores (morfológicos o moleculares) proveen información útil sobre algún nivel de la variación de los organismos. Lo importante es seleccionar el marcador apropiado para el nivel de variación de interés particular.
Por ejemplo, a nivel individual, los métodos más utilizados para detectar marcadores moleculares son: los "RFLPs", los "RAPDs", los "SSCP" y la "DGGE". También se ha usado la electroforesis de isoenzimas, los mini y microsatélites y las reacciones inmunológicas. En cambio, a nivel poblacional los más ampliamente utilizados son la electroforesis de isoenzimas, los "RFLPs", los "RAPDs", los mini y microsatélites y los "AFLPs". La hibridación de ADN también ha sido usada a nivel de poblaciones. Sin embargo, su mayor aplicación ha sido en estudios taxonómicos que involucran niveles taxonómicos más inclusivos como familias y órdenes.
Los métodos usados a nivel específico son los ensayos inmunológicos, la secuencia de aminoácidos, la electroforesis de isoenzimas, la hibridación de ADN, los "RFLPs" y las secuencias de ADN. Actualmente, los estudios comparativos de variación basados en las secuencias de ADN son muy utilizados sobre todo en el área de la sistemática. Sin embargo, estos trabajos están limitados, por ahora, a una región pequeña del genoma (usualmente un gen, un intrón, una secuencia espaciadora o regiones intergénicas). No obstante, el continuo desarrollo tecnológico está haciendo posible el muestreo de un número cada vez mayor de genes y taxa.
Desde la década pasada, muchos grupos de investigación interesados en problemas ecológicos y sistemáticos han incorporado en sus trabajos por lo menos alguno de los métodos disponibles para generar marcadores moleculares. Con los avances de la tecnología (equipo especializado, mejores reactivos, etc.) cada vez es más fácil y rápido la obtención de muchos de estos marcadores. No es difícil anticipar que se continuarán usando estos marcadores y que sin lugar a duda se desarrollarán nuevas metodologías para obtenerlos. Sin embargo, es indispensable seleccionar el marcador apropiado para un problema particular.
Por ejemplo para la mayoría de estudios a nivel intraespecífico la electroforesis de isoenzimas y las enzimas de restricción, particularmente los "RFLPs", estarían entre las técnicas a escoger pues a menudo se necesita examinar un gran número de individuos sobre un gran número de loci, por lo tanto, escoger una técnica también dependerá de la resolución requerida para el nivel taxonómico de interés. Además, se debe tener presente que un marcador molecular es útil cuando presenta propiedades como polimorfismo, codominancia, frecuencia en el genoma, distribuido en todo el genoma y ser selectivamente neutro. También, debe ser reproducible, de ensayo rápido y sencillo y de fácil acceso. Actualmente, no existe algún marcador molecular que satisfaga todos estos criterios. Por lo tanto, para un estudio comparativo de la variación ya sea ecológico o taxonómico, es necesario seleccionar algún marcador que por lo menos combine algunas de estas propiedades.

